AKTUELLE PROJEKTE
Der Forschungsverbund Leibniz INFECTIONS konzentriert sich auch weiterhin auf antimikrobielle Resistenzen (AMR). Die Ausbreitung von AMR bezeichnet die Weltgesundheitsorganisation (WHO) als eine der größten Bedrohungen für die öffentliche Gesundheit. Um das Problem ergebnisorientiert und strategisch vorausschauend anzugehen, ist ein multidisziplinärer und interdisziplinärer Ansatz erforderlich. Resistente Mikroorganismen entstehen und verbreiten sich nicht nur durch den Selektionsdruck antimikrobieller Therapien, sondern hängen auch mit menschlichen Aktivitäten zusammen. Dazu gehören:
- Einflüsse auf die Umwelt
- die Art der Landwirtschaft
- die öffentliche Gesundheit
- und die wirtschaftlichen und sozialen Bedingungen, die bestimmen, wie Medikamente entwickelt, verkauft und benutzt werden.
Um zu verstehen, warum Resistenzen entstehen und wie sie sich verbreiten, müssen Expertinnen und Experten aus vielen Fachrichtungen eng zusammenarbeiten – nicht nur aus der Medizin. Dementsprechend nutzt Leibniz INFECTIONS das Fachwissen von 15 Leibniz-Instituten und weiteren Partnern unter anderem dem Robert-Koch-Institut (RKI) und dem Friedrich-Loeffler-Institut (FLI).
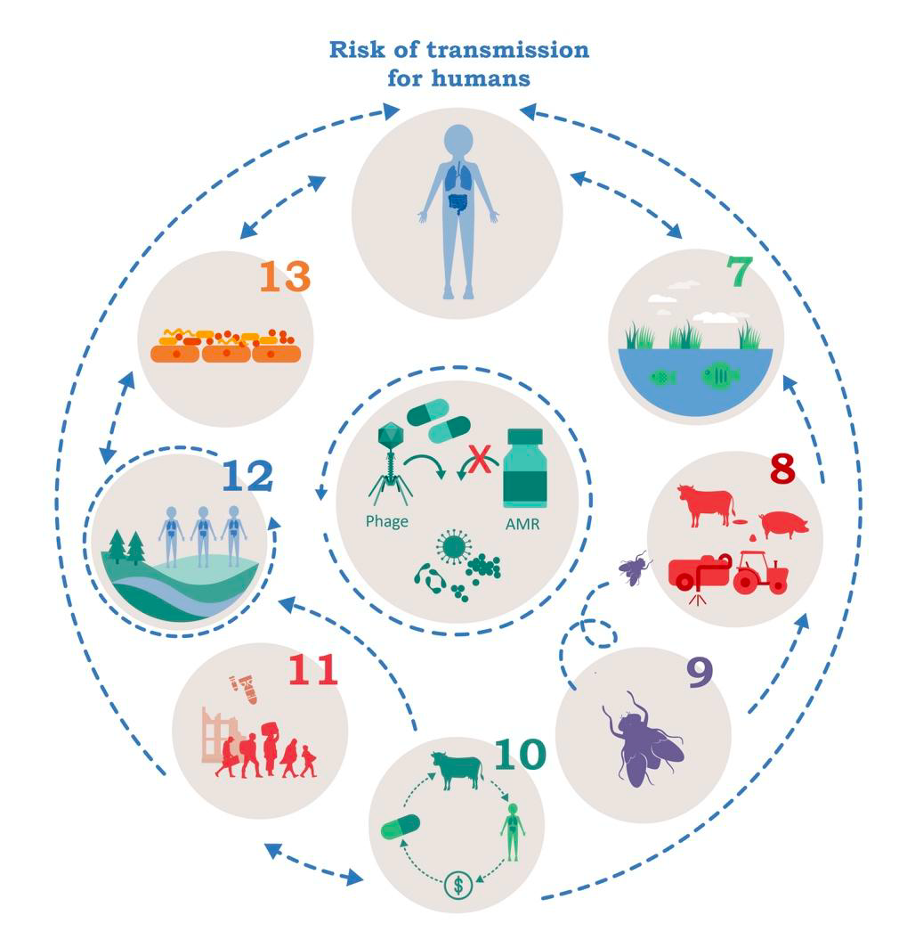
Es wurden sieben Forschungsprojekte konzipiert, in denen verschiedene Verbundpartner zusammenarbeiten. Die interdisziplinären Projektteams (IPT) untersuchen die Übertragung von AMR aus verschiedenen Quellen – nämlich menschlichen, ökologischen, landwirtschaftlichen und sozioökonomischen. Ziel ist es, die allgemeinen Treiber von AMR zu identifizieren, das Risiko der Übertragung auf die menschliche Gesundheit zu bewerten und Gegenstrategien zu entwickeln. Die IPTs sind fortlaufend von 7 bis 13 nummeriert, um sie von den IPTs 1 bis 6 der vorherigen Förderperiode zu unterscheiden (siehe Abbildung). Weitere Informationen stehen unten in den Beschreibungen der Projekte.
Unser zentrales Ziel ist es, die Ausbreitung von Antibiotika-resistenten Keimen zwischen Nutztieren, der Umwelt und dem Menschen zu verstehen. Dazu dient das IPT1 innerhalb des Leibniz-Forschungsverbunds INFECTIONS als zentraler Knotenpunkt für die bakteriologische, molekularbiologische und spektroskopische Identifizierung resistenter Bakterien sowie für bioinformatische Analysen umfangreicher, genomischer Sequenzierungsdaten. Die metagenomische Sequenzierung von DNA aus Proben, die in den anderen IPTs gesammelt wurden, wird kultivierungsunabhängige Informationen über die Vielfalt und Häufigkeit Antibiotika-resistenter Bakterien liefern. Wir werden Short-read- und Long-read DNA-Sequenzierung, spezifische Bioinformatik-Pipelines und öffentliche Datenbanken kombinieren, um Antibiotikaresistenzgene und -mutationen zu identifizieren. Anschließend werden besonders interessante Resistenzen (z. B. verbreitete Resistenzgene) in einer größeren Anzahl von Proben mit Hilfe der quantitativen PCR nachverfolgt. In selektiven Kultivierungsmedien werden Antibiotika-resistente Krankheitserreger aus Umweltproben isoliert. Die Bakterienisolate werden auf ihre Antibiotikaempfindlichkeit getestet und ihre Zellreaktionen auf Antibiotika werden mit Hilfe der Raman-Spektroskopie charakterisiert. Außerdem werden die Genome ausgewählter Bakterienisolate sequenziert, um die Quellen von Antibiotikaresistenz in der Umwelt zu ermitteln, z. B. aus Fliegen (IPT4), Hygienestudien (IPT5) und offenen Gewässern (IPT6).
Teilnehmende Institute: DSMZ, IPHT, IGB, IÖR, IZW
Die Entwicklung antimikrobieller Resistenzen (AMR) wird zu einem immer dringlicheren Problem in unserer Welt. Da immer mehr Mikroorganismen eine Resistenz gegen antimikrobielle Substanzen entwickeln, wird es immer schwieriger, Infektionskrankheiten zu bekämpfen, die bisher gut behandelbar waren. Im Rahmen des Interdisziplinären Projektteams 2 (IPT2) konzentrieren wir uns in erster Linie auf Mikroben, die Infektionen der Lunge verursachen können und von denen bekannt ist, dass sie Resistenzen gegen antimikrobielle Substanzen entwickeln können. Diese Infektionen treten in der Regel nicht bei gesunden Menschen auf, können aber bei Menschen mit abgeschwächtem Immunsystem oder Lungenkrankheiten wie Mukoviszidose vorkommen. Häufig verursachen diese Mikroorganismen nicht allein Infektionen, sondern bilden hochstrukturierte polymikrobielle Gemeinschaften, die als "Biofilme" bekannt sind. Angesichts der hohen Zelldichten und der Artenvielfalt ist es nicht verwunderlich, dass physikalische und soziale Wechselwirkungen innerhalb gemischter Biofilme zu Kooperation oder Wettbewerb zwischen den Zellen führen können, was evolutionäre Veränderungen von Eigenschaften der Mitglieder einer Biofilmgemeinschaft zur Folge haben kann, einschließlich der Entwicklung einer antimikrobiellen Resistenz, um nur eine mögliche Konsequenz aus einer mikrobiellen Evolution zu nennen. Angesichts der rasanten Entwicklung von Antibiotikaresistenzen und der Folgen für die Behandlung von Infektionskrankheiten ist es daher äußerst wichtig zu verstehen, wie sich die Mikroorganismen in gemischten Biofilm-bildenden Konsortien weiterentwickeln und an neue Umgebungen anpassen.
Im Rahmen des aktuellen Vorhabens wollen wir die Wechselwirkungen zwischen pulmonalen Opportunisten und auftretenden antimikrobiellen Resistenzen in gemischten Biofilmen untersuchen, wobei wir einen besonderen Schwerpunkt auf bakterielle und mykotische Erreger legen, die eine bedeutende Rolle bei Mukoviszidose-Patienten spielen, wie z.B. Stenotrophomonas maltophilia und Candida albicans. Um die Bedingungen des menschlichen Atmungssystems nachzuahmen und die Komplexität der in vivo Situation in einem in vitro Modell zu erreichen, werden wir die Biofilmgemeinschaften in physiologisch relevanten Zellkultursystemen an der Luft-/Flüssigkeits-Grenzfläche untersuchen.
Teilnehmende Institute: FZB, HKI, DPZ, DSMZ, LIV, IPHT, ISAS
Der zunehmende Einsatz von Antibiotika im Gesundheitswesen und in der Landwirtschaft treibt die antimikrobielle Resistenz voran. Fehlende Regularien und Kontrollmechanismen auf lokaler, nationaler und internationaler Ebene führen dazu, dass einige Länder bereits alarmierenden Raten antimikrobiell resistenter Krankheitserreger aufweisen. Eine Entwicklung mit schwerwiegenden Folgen für die Weltbevölkerung. Experten schätzen, dass bis 2050 jedes Jahr zehn Millionen Menschen an gewöhnlichen Infektionen sterben werden, die dann nicht mehr behandelbar sind. Das zentrale Ziel dieses Forschungsprojekts ist es, einen weltweit optimalen Einsatz von Antibiotika zu unterstützen. Der Fokus dieses Projekts liegt insbesondere auf zwei Bereichen: Zum einen der Verringerung des Einsatzes von Antibiotika, in Bereichen in denen diese ineffizient sind und zum anderen in der Förderung eines gleichberechtigten Zugangs zu hochwertigen Antibiotika. Fehlende Aufsichts- und Kontrollmechanismen sowie fehlende Tests und Diagnostik führen zu einem erhöhten Einsatz von Antibiotika, weit über das sozial verträgliche Niveau hinaus. Daher wird diese Projekt Maßnahmen zur besserer Diagnostik, und die Rolle von Informationen und Transparenz für die Eindämmung der Antibiotikaresistenz bewerten. Im Rahmen des Projektes werden Interventionen auf Mikro- und Makroebene ausgewertet und deren Kosten und Nutzen gegenübergestellt und politische Handlungsempfehlungen gegeben.
Teilnehmende Institute: BNITM, GIGA, FZB, IfW, RWI
In dem Projekt IPT4 geht es um Vektorökologie. Vektoren wie Fliegen sind in der Lage, verschiedene Pathogene wie zum Beispiel Bakterien, Viren, Pilze und Parasiten zu verbreiten. Die Hausfliege (Musca domestica) kann Bakterien über eine Distanz von 5-7 km transportieren. Sie ernährt sich von verrottendem organischem Material und Tierexkrementen und legt dort auch ihre Eier ab. Aufgrund ihrer koprophagischen Lebensweise nimmt sie Bakterien auf und ist in der Lage, diese später durch Regurgitation und Defäkation auf menschliche Lebensmittel zu übertragen. Durch den häufigen und präventiven Gebrauch von Antibiotika in der Tierhaltung kommt es zur Entstehung von antimikrobiell resistenten Bakterien. Diese Bakterien werden von den Fliegen aufgenommen und können von Tierhaltungsanlagen in den urbanen Raum eingetragen werden, wo sie schwere nosokomiale Infektionen auslösen können. Um zu verstehen, wie Fliegen die Bakterien zwischen verschiedenen Landschaftsstrukturen hin und her tragen, wird ein „Mark-Release-Recapture“ Versuch initiiert. Dafür werden adulte Fliegen (Musca domestica) gefangen, mit Farben markiert und wieder freigelassen. Anschließend wird die Wiederfangrate bestimmt. Diese Versuchsansatz soll helfen, die Flugradien und Habitatbindung der Hausfliege besser zu verstehen. Außerdem sollen Strategien ermittelt werden, die die Ausbreitung von antimikrobiell resistenten Bakterien behindern. Weitergehend soll in einem Laborversuch untersucht werden, ob sich die Bakterien in der Fliege vervielfältigen. Die drei Larvenstadien der Hausfliege entwickeln sich in den Tierexkrementen und nehmen dabei Bakterien auf. Von besonderem Interesse ist es, ob diese Bakterien die Metamorphose des Insekts überleben. Das Augenmerk liegt vor allem darauf, ob die antimikrobiellen Bakterien nach der Histolyse eher in den Puparien zu finden sind oder in den frisch geschlüpften Imagines.
Insgesamt soll dies Forschungsprojekt also dazu beitragen, zu verstehen, wie antimikrobiell resistente Bakterien die verschiedenen Stadien des Lebenszyklus der Hausfliege überstehen und wie diese von der Fliege verbreitet werden.
Teilnehmende Institute: ZALF, DSMZ, ATB, IÖR
Im Rahmen des Interdisziplinären Projekt Teams 5 (IPT5) arbeiten das ATB und seine Partner intensiv zusammen, um die Übertragungsmechanismen von antimikrobiellen Resistenzen (AMR) in der Nutztierhaltung zu ermitteln und mögliche Interventionsmaßnahmen abzuleiten. Wir vergleichen das Auftreten von AMR bei typischen konventionell gehaltenen Schweinen in einer standardmäßigen und einer verbesserten hygienischen Umgebung (durch den Einsatz von Insektiziden, Fliegenfallen, verstärkter Desinfektion und Staubreduktion). Darüber hinaus wird der antibakterielle Effekt verschiedener Futterzusätze im Vergleich zum Standardfuttermittel ohne Zusätze verglichen, basierend auf der mikrobiellen Besiedlung im Darm von Ferkeln während der frühen Mastperiode. In enger Zusammenarbeit mit anderen INFECTIONS-Partnern wird das Überleben und die Vermehrung von AMR-Mikroben im Schweinekot, Staub und Fliegen unter allen Bedingungen analysiert. Die Häufigkeit von AMR wird anhand von bakteriologischen Methoden, PCR und kultivierungsunabhängiger DNA-Sequenzierung ermittelt. Mit Unterstützung anderer IPTs werden die Ergebnisse genutzt, um Maßnahmen und potenzielle Strategien zur Eindämmung der Ausbreitung von AMR in der kommerziellen Tierhaltung zu definieren. Dadurch soll die potenzielle Kontamination der Umwelt (z. B. von Oberflächengewässern bei Verwendung kontaminierter Gülle als organischer Dünger) reduziert werden.
Teilnehmende Institute: ATB, DSMZ, ZALF, HKI, IPHT, IÖR, VRC
Die Weltgesundheitsorganisation (World Health Organization, WHO) hat die antimikrobielle Resistenz (AMR) zu einer der zehn größten Bedrohungen für die öffentliche Gesundheit in der Gegenwart und in naher Zukunft erklärt. Laut WHO wird AMR innerhalb einer Generation katastrophale Auswirkungen haben, wenn nicht endlich ein dringender und wirksamer globaler Plan zur Eindämmung umgesetzt wird. Nach Schätzungen einer aktuellen Studie basierend auf Daten aus 204 Ländern standen 2019 1,27 Millionen Todesfälle im Zusammenhang mit AMR-Bakterien. Eine der Herausforderungen bei der Überwachung von AMR-Organismen und antimikrobiellen Resistenzgenen (ARGs) ist ihre ubiquitäre Verbreitung, d. h. sie kommen in der Regel in Menschen, Tieren, Pflanzen, Mikroben und in der Umwelt wie Wasser, Boden und Luft vor. Insbesondere Wasser spielt eine Schlüsselrolle als Vektor und Reservoir für die Übertragung von AMR in städtischen und ländlichen Gebieten. Unsere vorläufigen Ergebnisse deuten darauf hin, dass die Häufigkeit von AMR in städtischen Gewässern und Sedimenten im Vergleich zu ländlichen Gewässern wesentlich höher ist. Darüber hinaus wird beobachtet, dass Sedimente in ländlichen Seen und Wasser aus landwirtschaftlichen Flächen aufgrund des häufigen Einsatzes von Antibiotika in der Viehzucht eine wichtige Quelle für AMR in der Umwelt darstellen. IPT5 zielt darauf ab, weitere Erkenntnisse über die Ausbreitung von AMR aus Kläranlagen in die Umwelt zu gewinnen und die Humanisierung des Mikrobioms von städtischem Wasser zu charakterisieren. Insbesondere die Verwendung von Long-Read-Sequenzierung in diesem Projekt wird es ermöglichen, mobile genetische Elemente (MGEs) wie Plasmide und genomische Inseln (GIs) und damit eine höhere Auflösung in der Metagenomanalyse zu erzielen. Außerdem wird eine Risikobewertung durchgeführt, um die Umweltexposition gegenüber ARGs zu beurteilen. Die Ergebnisse aus diesem Projekt werden es ermöglichen, die Häufigkeit und Diversität von AMR und den sie tragenden Bakterien direkt mit anderen Erkenntnissen zu verknüpfen und in Zusammenarbeit mit weiteren Verbundpartnern des Leibniz-Forschungsverbunds INFECTIONS einen breiten Überblick über AMR-Profile in verschiedenen Gewässern und potenziellen Krankheitsüberträgern in der Region Berlin-Brandenburg zu geben.
Teilnehmende Institute: IZW, IGB, LIV, DPZ, GIGA, IÖR
Wasser ist für das menschliche Leben unverzichtbar, spielt aber auch eine wichtige Rolle als Überträger und Reservoir für die Übertragung von AMR in städtischen und ländlichen Gebieten. Unsere Ergebnisse deuten darauf hin, dass die Häufigkeit und Vielfalt von AMR in städtischen Gewässern und Sedimenten höher ist als in ländlichen Gewässern. Kläranlagen sind eine wichtige Quelle für AMR in städtischen Wasserquellen. Darüber hinaus sind Sedimente aus ländlichen Seen und Wasser aus landwirtschaftlichen Flächen wichtige Quellen für AMR in der Umwelt.
Wir werden das Risiko des Kontakts des Menschen mit AMR durch Umweltwasser bewerten, indem wir unsere in der vorherigen Förderperiode im Rahmen von INFECTIONS gesammelten Daten nutzen und einen bioinformatischen Vergleich mit Sequenzen klinischer Isolate aus unseren Sammlungen durchführen. Die Analysen werden auf Pilzpathogene, einschließlich mobiler genetischer Elemente, ausgeweitet. Die zukünftige Wasserentnahme wird Abwasserproben aus Partnerländern umfassen, in denen Antibiotika/Antimykotika in der Landwirtschaft eingesetzt werden. Der Phagengehalt der Proben wird bestimmt. Wir untersuchen das Phagenom (alle Bakteriophagen im Wasser) und ob Phagen vielversprechende Alternativen zur Bekämpfung von AMR-tragenden Bakterien sein könnten. In Pilotstudien werden wir versuchen, AMR-haltige Bakterien aus Abwasser zu entfernen, indem wir in Laborstudien umweltbasierte Phagen einsetzen.
Teilnehmende Institute: ATB, BNITM, DSMZ, FZB, HKI, IGB, IZW
Angesichts des stetig steigenden weltweiten Fleischkonsums und der wachsenden Zahl von Nutztieren stellen durch Wirtschaftsdünger verbreitete Antibiotikaresistenzen (AMR) ein zunehmendes Problem dar, das die Gesundheit von Tieren, Menschen und Umwelt beeinträchtigt. Da Gülle im Sinne einer nachhaltigen Kreislaufwirtschaft als Dünger für landwirtschaftliche Felder genutzt wird, besteht eine potentielle AMR-Übertragungsquelle in die Nahrungskette des Menschen. Im interdisziplinären Projektteam 8 (IPT8) konzentrieren wir uns auf einen One-Health-Ansatz um die Dynamik von Mikroorganismen und Fliegenlarven in Güllebehältern zu untersuchen. Dabei werden verschiedene Güllelagerbedingungen in landwirtschaftlichen Betrieben und im Labor untersucht, wobei der Schwerpunkt auf der Persistenz von AMR-Bakterien liegt, darunter Krankheitserreger wie Salmonellen, ESBL-produzierende E. coli und nicht-tuberkulöse Mykobakterien. Unser Ziel ist es, das Vorkommen von AMR und Veränderungen des Mikrobioms in Güllelagerbehältern in landwirtschaftlichen Betrieben und im Labor zu überwachen. Der Einfluss saisonaler Temperaturschwankungen, der Gülleart und des Füllstands der Behälter wird untersucht. Wir werden AMR-Krankheitserreger in Gülle durch mikrobiologische Kultivierung nachweisen und quantifizieren. Zur Bewertung der Mikrobiomvielfalt in Gülle wird eine 16S-rDNA-Sequenzierung durchgeführt. Es werden Untersuchungen durchgeführt um das Vorkommen und die Aufnahme von AMR-Bakterien aus Gülle durch Fliegen zu untersuchen. Die Pathogenbelastung von Fliegen und ihren Larven wird mit bakteriologischen und molekularen Verfahren bestimmt. Die hier gewonnenen Forschungsergebnisse ermöglichen es, praktische und wirksame Maßnahmen zur Reduzierung von AMR-Bakterien abzuleiten, indem die optimalen Bedingungen zur Güllelagerung ermittelt werden.
Teilnehmende Institute: ATB, BNITM, DSMZ, FZB, IGB, IZW, ZALF
Das interdisziplinäre Projektteam IPT9 untersucht, wie Fliegen antimikrobiell resistente (AMR) Krankheitserreger von Nutztieren auf den Menschen übertragen können, und bewertet die daraus resultierenden Gesundheitsrisiken. Die Studie konzentriert sich auf die Stubenfliege (Musca domestica), schließt aber auch andere Fliegenarten ein, die in der Nähe von Viehbeständen vorkommen. In Feldexperimenten werden wir in Deutschland und Ghana Fliegenarten, Populationsdichten, Bewegungsmuster und die Belastung mit Krankheitserregern bestimmen. In Laborstudien wird untersucht, wie Fliegen AMR-Erreger wie multiresistente Enterobakterien, Vancomycin-resistente Enterokokken, nicht-tuberkulöse Mykobakterien und Candida-Arten aufnehmen und ausscheiden. Auf der Grundlage ökologischer Daten und experimenteller Erkenntnisse werden Modelle entwickelt, um die Ausbreitung und Übertragung von Krankheitserregern durch Fliegen vorherzusagen und das Risiko für den Menschen zu bewerten. Das Team wird auch untersuchen, wie Umweltfaktoren die Übertragungsdynamik beeinflussen und die Wirksamkeit verschiedener Bekämpfungsstrategien untersuchen. Mehrere Leibniz-Institute bringen ihre Expertise in den Bereichen Entomologie, Mikrobiologie, Ökologie und Risikoanalyse ein, um ein umfassendes Verständnis der durch Fliegen vermittelten AMR-Verbreitung zu erlangen.
Teilnehmende Institute: ATB, BNITM, DSMZ, FLI, FZB, HKI, IOER, ZALF
Der unregulierte und massenhafte Einsatz von Antibiotika in der Tierhaltung trägt maßgeblich zur weltweiten Ausbreitung von Antibiotikaresistenzen bei. In vielen Regionen des Globalen Südens fehlt es an tierärztlicher Versorgung und an regulatorischer Kontrolle, wodurch Antibiotika oft ohne fachgerechte Aufsicht verabreicht werden. Ziel von IPT10 ist es, praxistaugliche Interventionen zur Eindämmung von AMR in ressourcenarmen Kontexten zu entwickeln und zu testen.
Dazu kombinieren wir drei Ansätze: (1) Verhaltensänderung durch Informationskampagnen, (2) der Einsatz kostengünstiger molekularer Diagnostik für Tierkrankheiten und (3) die digitale Erfassung von Antibiotikaeinsatz und Resistenzdaten. In Kooperation mit Partnerinstitutionen in Burundi, Ruanda, Tansania, Ghana und der DR Kongo werden diese Interventionen unter realen Bedingungen getestet.
Die Grundlage bilden bereits laufende Projekte wie AMRAfrica zur digitalen Überwachung, ADA zur Entwicklung neuer Diagnostik sowie mobile AMR-Labore in Ostafrika. IPT10 verbindet diese innovativen Ansätze mit empirischer Forschung, um praxistaugliche Empfehlungen für nachhaltige AMR-Bekämpfung zu entwickeln.
Teilnehmende Institute: ATB, BNITM, IPHT, RWI
Teilnehmende Institute: BNITM, FZB, IfW, RKI, RWI
Nicht-tuberkulöse Mykobakterien (NTM) sind eine vielfältige Gruppe von Bakterien, die häufig in der Umwelt vorkommen, insbesondere in Wasser und Boden. Sie verursachen zunehmend Infektionen beim Menschen, insbesondere bei Personen mit geschwächtem Immunsystem oder vorbestehenden Lungenerkrankungen wie Mukoviszidose, und es ist bekannt, dass sie auch eine Vielzahl von Tierarten infizieren. Diese Infektionen sind oft schwer zu behandeln, da viele NTM von Natur aus gegen mehrere Antibiotika resistent sind.
Es wird angenommen, dass Menschen sich hauptsächlich durch Kontakt mit Umweltquellen, die diese Bakterien enthalten, mit NTM infizieren. Welche Umweltquellen derzeit das höchste Risiko für anfällige Personen darstellen, ist jedoch noch unbekannt.
Ziel dieses Projekts ist es, NTM aus verschiedenen Umweltquellen wie Boden und Wasser sowie aus Pflanzen und Tieren in ganz Deutschland zu sammeln und zu analysieren. Die Bakterien werden untersucht, um festzustellen, wie häufig sie vorkommen, wie genetisch vielfältig sie sind und ob sie gegen die Antibiotika resistent sind, die normalerweise zur Behandlung von NTM-Erkrankungen eingesetzt werden. Diese Umwelt-NTM werden auch mit klinischen Isolaten von menschlichen Patienten unter Verwendung moderner molekularer Werkzeuge verglichen.
Die Ergebnisse werden uns helfen, potenzielle Quellen und Übertragungswege von NTM besser zu verstehen, Infektionsrisiken zu bewerten und zur Entwicklung neuer Strategien für Prävention und Behandlung beizutragen. Vielversprechende neue Therapien, darunter Bakteriophagen und neuartige Wirkstoffe, werden in Zusammenarbeit mit dem Projekt IPT13 an diesen Stämmen getestet.
Teilnehmende Institute: ATB, DSMZ, FZB, FLI, IGB, IZW, NTUS, RKI, UoS
In IPT13 werden die komplexen Wechselwirkungen polymikrobieller Spezies weiter untersucht. Auch hier werden Lungenepithelzellen der Luft-Flüssigkeits-Grenzfläche als Modelloberfläche für die Kultivierung des Biofilms verwendet. In der nächsten Phase werden Lungenorganoide etabliert, um ein komplexeres Wachstumssystem zu schaffen. Es werden drei Gruppen von Mikroorganismen untersucht: Bakterien – M. abscessus, S. maltophilia, Pilze: C. albicans, C. auris, A. fumigatus und Viren: Influenza und SARS-CoV.
Die Ziele dieses Projekts bestehen darin, die Wechselwirkungen dieser Mikroorganismenarten mittels Lebendzellmikroskopie zu bewerten, mittels Proteomik zu analysieren, wie die einzelnen Arten in einem gemischten Biofilm aufeinander reagieren, und Phagenisolate und neuartige Antibiotika gegen polymikrobielle Biofilme für die therapeutische Kontrolle zu untersuchen.

